填补AlphaFold3空白,字节跳动提出物理引导的方法让蛋白质动起来
文章摘要
【关 键 词】 生物分子、蛋白质结构、动态预测、药物开发、深度学习
在探索蛋白质等生物分子功能的过程中,从预测静态结构到动态构象分布的跨越显得至关重要。这不仅能帮助理解生物过程,还能促进药物位点的识别和抑制剂的设计。然而,传统的分子动力学模拟方法在时间尺度的跨越上存在局限,而当前的深度学习模型如AlphaFold 3也主要集中于静态结构的预测。蛋白质构象实际上遵循玻尔兹曼分布,现有研究在采样和质量上仍未能充分模拟这一分布。
来自字节跳动的研究人员提出了CONFDIFF模型,这是一种物理信息引导的蛋白质构象生成扩散模型。通过预测中间时刻的力场与能量,模型能够生成高质量且低能的构象,更好地符合真实的玻尔兹曼分布。这一创新方法绕开了实时能量和力场计算的昂贵成本,并通过实验证明了其有效性。
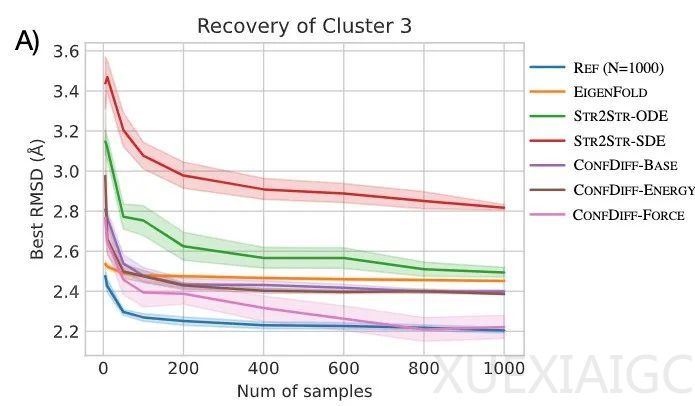
在评估中,CONFDIFF在快速折叠蛋白质和牛胰蛋白酶抑制剂的构象生成任务上表现出色。模型不仅提高了构象的生成有效性,保持了多样性,而且在预测不同亚稳态方面也展现了更好的能力。特别是在力场引导下,模型采样效率更高,生成的构象更加稳定。
这项研究不仅推进了蛋白质结构预测从静态到动态的进程,也为药物开发提供了有力工具。字节跳动AI制药团队在生成式蛋白质设计、蛋白质构象预测及冷冻电镜解析等多个领域取得了显著成果,其工作在顶级学术会议上得到了认可,为揭示蛋白质机理和探索生命本质贡献了重要力量。
原文和模型
【原文链接】 阅读原文 [ 3183字 | 13分钟 ]
【原文作者】 机器之心
【摘要模型】 glm-4
【摘要评分】 ★★★★★
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章
暂无评论...