文章摘要
【关 键 词】 生物医学、专业术语、Mamba架构、问答任务、文本分类
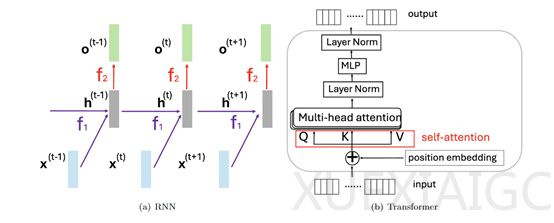
生物医学文献的复杂性对传统模型构成挑战,因为它们难以处理专业术语的多义性和快速更新的知识。为了应对这些挑战,美国伦斯勒理工学院和斯坦福大学医学院的研究人员开发了BioMamb,这是一个基于Mamba架构的专业分析大模型。Mamba架构通过结构化状态空间模型和将参数作为输入函数,实现了线性时间复杂度,有效解决了传统Transformer模型在处理长序列时的效率问题。
BioMamb的开发包括预训练和微调两个阶段。在预训练阶段,模型使用Mamba-130m模型的权重初始化,并在PubMed摘要等生物医学文本上进一步训练。预训练采用自回归方法,使模型能够从左至右理解文本流,适合生成连贯文本。微调阶段,BioMamb在BioASQ事实数据集上进行监督学习,优化问答任务的性能。
BioMamb在多个生物医学NLP任务上表现出色,包括问答、文本分类和实体识别等,其准确率和精确率显著优于现有模型。这表明BioMamb能有效处理生物医学语言的细微差别,提供准确的答案。BioMamb的开发为生物医学文献的分析提供了一个强大的工具,有助于推动该领域的研究和应用。
原文和模型
【原文链接】 阅读原文 [ 1055字 | 5分钟 ]
【原文作者】 AIGC开放社区
【摘要模型】 moonshot-v1-32k
【摘要评分】 ★☆☆☆☆
© 版权声明
文章版权归作者所有,未经允许请勿转载。
相关文章
暂无评论...




